Na Parte 1 desse post (que já publiquei faz um tempão!) eu fiz uma classificação de imagens de dígitos escritos a mão usando o k-nn (algoritmo dos vizinhos mais próximos) usando as informações das imagens sem nenhum tipo de tratamento, isto é, sem nenhum método de preparação. Como foi mostrado, o k-nn só foi capaz de classificar razoavelmente bem com com k = 1 e conseguiu uma acurácia de apenas 78%, algo muito distante do que ainda pode ser conseguido.
Na Parte 2 eu trabalhei com um método de redução de dimensionalidade, o PCA, e também foram explorados diversos outros classificadores, como o Random Forest, o SVM e etc. O resumo dos resultados foi o seguinte:
- k-nn com k = 1: 84%
- Regressão linear: 14%
- Regressão Logística Multinomial: 64%
- Árvores de decisão: 24%
- RandomForest: 72%
E a conclusão geral foi que não foi possível bater o k-NN ou ainda mais chegar aos resultados reportados na literatura, superiores a 95% de acurácia na tarefa. Como foi mencionado anteriormente, os possíveis problemas foram:
- Conjunto pequeno de imagens.
- Modelos com parâmetros default.
Assim, nessa última parte vou mostrar como é possível treinar melhores modelos com muito mais dados e como é possível melhorar a performance dos algoritmos com melhores hiperparâmetros. Outro ponto importante que eu queria mostrar também é o uso do pacote caret para automatizar diversas tarefas desse processo.
1.Dados e visualização
Como eu comentei antes, vamos nessa parte tentar utilizar um conjunto maior de imagens. Para tanto, ao invés desse pequeno conjunto de teste e treino que foi fornecido na Parte 1, vamos utilizar um conjunto muito maior de images de dígitos escritos a mão, o famoso data set MNIST. Se você verificar nesse link, você vai encontrar diversas informações com respeito a esse conjunto de dados e também você pode baixa-lo e utilizar nas análises que se seguem, caso queira reproduzir o que você está vendo aqui. ENTRETANTO, para poupar o seu trabalho e o meu, ao invés de pegar os dados diretamente deste site, vamos utilizar esse mesmo conjunto de dados já tratado e preparado pela equipe do Kaggle. A vantagem é que o arquivos já estão no formato csv e não será mais necessária a etapa de preparação realizada na Parte 1 e na Parte 2 desta série. Outra vantagem é que após você rodar estes modelos, se você quiser, você pode submeter seus resultados no Leaderboard para experimentar como funciona esse site de competição.
Na Parte 2, como eu peguei diretamente as imagens e converti em uma matriz, agora você poderia ficar confuso e se perguntar: mas e aí, como são estas imagens? como eu vou ver se já está em csv? Para você não ficar com dúvida, vamos “imprimir” as imagens.
############ Explorando as imagens ###############################
## Contando o número de imagens por dígito
barplot(table(treino$label), ylim = c(0,5000))
## Transformando em uma matriz
treino <- as.matrix(treino)
## Imprimindo uma imagem
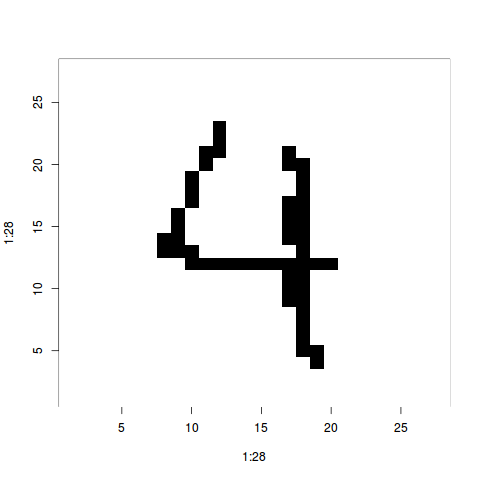
matriz_imagem <- matrix(treino[1000,-1], ncol = 28)
matriz_imagem <- matriz_imagem[,28:1] ## invertendo a imagem
image(1:28, 1:28, matriz_imagem, col = c('white', 'black'))
Veja que eu peguei a primeira linha do conjunto de treino, transformei em uma matriz e imprimi a matriz como uma imagem. Nesse caso é o dígito 4. Assim, apesar de agora você estar usando um arquivo em csv preparado eles fizeram a mesma coisa que eu fiz anteriormente. Se você quiser entender melhor como cada imagem virou uma linha dessa tabela dá uma olhada na Parte 1 dessa série. Só para mostrar que os dígitos estão ok, eu vou imprimir uma “imagem média”, onde em cada imagem eu tenho um valor médio em cada píxel considerando todas as imagens do conjunto de dados.
## Plotando uma imagem média para cada dígito
## Definindo uma escala de cor, indo do branco ao preto
colors <- c('white','black')
cus_col <- colorRampPalette(colors=colors)
## Plot de cada imagem média
## Divindo a tela
png('todos_digitos.png')
par(mfrow=c(4,3),pty='s',mar=c(1,1,1,1),xaxt='n',yaxt='n')
## Criando um array para armazenar as matrizes de cada imagem média
all_img <- array(dim=c(10,28*28))
## Recuperando todas as imagens por dígito e calculando a média
for(di in 0:9) {
print(di)
all_img[di+1,] <- apply(treino[treino[,1]==di,-1],2,sum)
all_img[di+1,] <- all_img[di+1,]/max(all_img[di+1,])*255
z<-array(all_img[di+1,],dim=c(28,28))
z<-z[,28:1] ##right side up
image(1:28,1:28,z,main=di,col=cus_col(256))
}
Até há uma certa variação (por isso que há uma sombra) mas no geral os mesmos píxels tem uma intensidade maior considerando cada dígito diferente que foi escrito na imagem. Isso nos leva a crer que os modelos devem conseguir distinguir um dígito do outro.
2. Preparação com PCA
Depois que você baixar os arquivos train.csv e test.csv do Kaggle já podemos efetuar a leitura dos arquivos e a preparação por meio do PCA. O que vamos fazer é aplicar o PCA e retendo somente o número de componentes necessário para alcançar 95% da variância total. Os detalhes sobre isso eu discuti na Parte 2.
## Leitura dos conjuntos de dados de treino e de teste
treino = read.csv('train.csv', header = T)
teste = read.csv('test.csv', header = T)
############ APlicação do PCA ####################################
## Obtendo componentes principais
pc <- prcomp(treino[,-1])
treino_pc <- pc$x
## Obtendo as variâncias acumuladas
vars = pc$sdev^2
props = vars/sum(vars)
varAcum = cumsum(props)
which.min(varAcum < 0.90)
## Aplicando a rotação nos dados de teste
teste_pc <- predict(pc, newdata = teste)
## Salvando treino e teste com PCA
save(treino_pc, file = 'treino_pc.rda')
save(teste_pc, file = 'teste_pc.rda')
eu costumo salvar os arquivos após cada etapa de preparação de forma a não precisar realizar o processo posteriormente. Outro ponto importante é que salvando os objetos no formato nativo do R, caso você precise recarregar os dados, o processo é muito mais rápido que a leitura em csv.
3. Árvore de Decisão
Como uma primeira tentativa, vamos utilizar o algoritmo para árvores de decisão do pacote rpart. Vamos utilizar todas as PC’s e treinar a árvore em 3/4 dos dados. O teste será realizado no 1/4 que foi separado.
################################################################# ## Teste com árvore de decisão library(rpart) ## Separando o conjunto treino em dois para avaliação set.seed(1) inTrain <- createDataPartition(treino$label, p = 3/4, list = F) train <- treino_pc[inTrain,] evaluation <- treino_pc[-inTrain,] ## Data frame de treino e teste treino_arvore = as.data.frame(cbind(train[,1:784])) treino_arvore$classes = as.factor(treino$label[inTrain]) ## Conjunto de teste para avaliação teste_arvore = as.data.frame(evaluation[,1:784]) ## Criando uma árvore arvore <- rpart(classes ~ ., data = treino_arvore) ## Calculando a matriz de confusão confusionMatrix(predict(arvore, teste_arvore, type = 'class'), treino$label[-inTrain])
e o resultado da matriz de confusão:
Confusion Matrix and Statistics
Reference
Prediction 0 1 2 3 4 5 6 7 8 9
0 637 2 13 20 3 66 17 12 0 2
1 0 995 9 7 28 7 3 75 6 53
2 60 54 741 61 30 97 130 16 93 10
3 185 45 111 803 13 270 106 29 89 30
4 3 0 16 10 796 73 16 82 16 471
5 86 13 46 88 36 341 31 61 185 49
6 24 16 47 45 21 40 725 4 13 43
7 17 0 4 2 20 16 2 652 10 85
8 18 46 40 27 11 60 12 18 532 13
9 14 0 6 10 74 6 13 103 57 305
Overall Statistics
Accuracy : 0.6217
95% CI : (0.6124, 0.631)
No Information Rate : 0.1115
P-Value [Acc > NIR] : < 2.2e-16
Kappa : 0.5796
Mcnemar's Test P-Value : < 2.2e-16
mostra que o desempenho ainda está longe do satisfatório. Com uma acurácia global de apenas 62% estamos ainda muito longe da meta de 95%. Veja que utilizamos a estratégia holdout, e com relação a Parte 2 desta série a única mudança é o fato de estarmos trabalhando com imagens com mais resolução e um conjunto maior. Parece que isso ainda não é o suficiente, assim vamos explorar outros algoritmos e vamos utilizar métodos de validação cruzada para encontrar os melhores hiperparâmetros.
4. RandomForest
O randomforest é um dos algoritmos de machine learning mais utilizados na indústria. Seu sucesso advém do fato de ser robusto, facilmente paralelizável e apresentar um desempenho muito bom em uma grande quantidade de problemas diferentes. Assim, vamos experimentar esse classificador procurando ajustar os melhores hiperperâmetros por validação cruzada. No caso do RF temos que definir qual o melhor m, um parâmetro que determina quantas variáveis são sorteadas na escolha do split em cada nó, de cada árvore de decisão do comitê. Se não ficou claro para você o que significa este hiperparâmetro não tem problema, não é difícil encontrar material onde você pode entender os detalhes do RF. O importante aqui é você entender que o valor do hiperparâmetro será escolhido com base no próprio conjunto de dados, utilizando validação cruzada.
#################################################################
## Teste com RandomForest
library(randomForest)
## Separando o conjunto treino em dois para avaliação
set.seed(1)
inTrain <- createDataPartition(treino$label, p = 3/4, list = F)
train <- treino_pc[inTrain,]
evaluation <- treino_pc[-inTrain,]
## Data frame de treino e teste, aqui retendo somente 160 PC's, equivalente a 95% de ## variância.
treino_arvore = as.data.frame(cbind(train[,1:160]))
treino_arvore$classes = as.factor(treino$label[inTrain])
## Conjunto de teste para avaliação
teste_arvore = as.data.frame(evaluation[,1:160])
## Modelagem
fitControl <- trainControl(method = "oob", verboseIter = T,
## Estimate class probabilities
classProbs = F)
set.seed(825)
rfFit <- train(classes ~ ., data = treino_arvore, verbose = T,
method = "rf",
trControl = fitControl,
tuneLength = 8,
metric = "Accuracy")
save(rfFit, file = 'rfFit.rda')
## Calculando a matriz de confusão
confusionMatrix(predict(rfFit, teste_arvore, type = 'raw'), treino$label[-inTrain])
e a matriz de confusão:
Confusion Matrix and Statistics
Reference
Prediction 0 1 2 3 4 5 6 7 8 9
0 1048 0 3 0 1 1 1 0 0 1
1 0 1130 2 0 1 1 0 2 3 0
2 0 3 1021 10 4 2 1 6 2 0
3 0 1 5 1073 3 12 0 1 8 11
4 1 0 6 0 970 4 1 1 4 18
5 0 0 0 8 2 932 6 0 4 6
6 6 1 3 2 5 3 1023 1 2 0
7 1 1 4 4 0 0 0 1077 3 7
8 0 0 5 4 5 2 1 2 967 4
9 2 2 3 2 11 2 0 1 2 1020
Overall Statistics
Accuracy : 0.9774
95% CI : (0.9744, 0.9802)
No Information Rate : 0.1084
P-Value [Acc > NIR] : < 2.2e-16
Kappa : 0.9749
e enfim chegamos a 97%!
5. SVM (Máquina de vertores de suporte)
O SVM é um algoritmo do tipo “caixa preta”. O princípio por detrás do algoritmo é criar hiperplanos separadores em dimensões maiores do que as presentes no conjunto de dados. A ideia é que se os pontos são linearmente separáveis, isto é, se um hiperplano como fronteira de decisão conseguiria separar completamente as classes, então o SVM é um método que pode ser utilizado para encontrar esse hiperplano. ENTRETANTO, ocorre que muitos problemas não são linearmente separáveis, e ainda que fossem não valeria a pena usar o SVM. Quando o problema não é linearmente separável o SVM, de uma certa forma, projeta os dados em um espaço onde é possível criar um hiperplano separador. Também, ele aceita um certo grau de “impurezas” dentro das fronteiras de decisão. Enfim, é um algoritmo do tipo “caixa preta”, que não tem origem na estatística já que é basicamente um algoritmo de otimização. Mas o fato é que o SVM apresenta resultados muito bons e uma grande quantidade de problemas e vamos ver isso aqui nesse teste.
#################################################################
## Teste com SVM RBF
## Carregando os pacotes
library(caret)
## Modelagem
fitControl <- trainControl(method = "cv", verboseIter = T,
## Estimate class probabilities
classProbs = F)
set.seed(825)
svmFit <- train(classes ~ ., data = treino_arvore,
method = "svmRadial",
trControl = fitControl,
preProc = c("center", "scale"),
tuneLength = 8,
metric = "Accuracy")
save(svmFit, file = 'svmFit2.rda')
## Calculando a matriz de confusão
confusionMatrix(predict(svmFit, teste_arvore, type = 'raw'), treino$label[-inTrain])
e avaliando no conjunto de avaliação:
Confusion Matrix and Statistics
Reference
Prediction 0 1 2 3 4 5 6 7 8 9
0 1046 0 3 0 1 3 5 1 3 6
1 1 1128 1 2 2 0 1 3 2 1
2 0 3 1015 13 4 3 1 15 4 1
3 1 1 4 1058 0 13 0 1 7 9
4 2 2 10 1 972 1 4 10 1 21
5 0 1 2 12 0 924 7 0 3 5
6 5 0 2 1 5 10 1010 0 4 0
7 0 0 5 7 0 0 0 1051 0 13
8 3 2 9 4 0 2 5 1 967 3
9 0 1 1 5 18 3 0 9 4 1008
Overall Statistics
Accuracy : 0.9696
95% CI : (0.9661, 0.9728)
No Information Rate : 0.1084
P-Value [Acc > NIR] : < 2.2e-16
Kappa : 0.9662
e novamente conseguimos algo em torno de 97%. Para ser honesto, depois que eu criei os modelos com os melhores hiperparâmetros, submetendo no Kaggle o SVM supera os 98%. Foi o modelo com melhor desempenho nessa tarefa.
Conclusão
Acho que depois da Parte 1, Parte 2 e Parte 3 (esta aqui!) você viu como se trabalha com classificação de imagens, como se prepara esse tipo de dado e como é possível alcançar altíssima acurácia utilizando os modelos de machine learning que você encontra por aí. Se você for reproduzir os exemplos, fique ciente que a etapa de modelagem pode demorar muito. No meu caso alguns destes modelos demoraram mais de 8 horas para o ajuste com os melhore hiperparâmetros! Outro ponto que eu não abordei é como o caret seleciona os melhores hiperparâmetros por validação cruzada. Isso vou deixar para falar com maior detalhe em outra oportunidade. Também gostaria de salientar que uma modelagem como essa é algo tipicamente diferente do que se espera em uma análise estatística tradicional. Aqui não estávamos interessados em inferência, mas sim em produzir modelos com o maior poder preditivo possível. Em casos como esse, trabalhar com métodos “caixa preta” não é em si um problema. O ponto principal é ter certeza que seu modelo apresentará um bom desempenho no futuro, com novos dados. POR FIM, esses modelos não são o estado da arte nesta tarefa, já que com deep learning é possível passar dos 99% de acurácia.

[…] Nessa última parte da sequência sobre como criar modelos preditivos para predição de imagens de dígitos, vou mostrar como é possível treinar melhores modelos com muito mais dados e como é possível melhorar a performance dos algoritmos com melhores hiperparâmetros. Referência: rmining.net […]